Mutation Matrix レポート¶
ここでは、サンプルデータ [*] を使用して、Mutation Matrix レポートを出力するために必要な入力データと設定方法を解説します。
| [*] | サンプルデータは paplot をダウンロードして解凍したディレクトリ中、example ディレクトリにあります。 |
1. 最小データセット¶
paplot で Mutation Matrix を作成するために最低限必要な項目はサンプル名 (Sample)、遺伝子名 (Gene)、変異タイプ (MutationType) の3つです。
Sample,MutationType,Gene
SAMPLE00,intronic,GATA3
SAMPLE00,UTR3,CDH1
SAMPLE00,exonic,GATA3
SAMPLE01,splicing,WASF3
SAMPLE01,intronic,WASF3
SAMPLE01,exonic,NRAS
SAMPLE02,intronic,FBXW7
SAMPLE02,intronic,GATA3
SAMPLE02,ncRNA_intronic,ACVR2B
SAMPLE03,exonic,CAP2
SAMPLE03,intronic,PIK3CA
SAMPLE03,downstream,SEPT12
今回の例では列名を Sample, MutationType, Gene としていますが、任意に設定できます。
設定ファイルの [result_format_mutation] セクションでデータの列名を次のように設定します。
[result_format_mutation]
col_group = MutationType
col_gene = Gene
col_opt_id = Sample
編集した設定ファイルを使用して paplot を実行します。
paplot mutation {unzip_path}/example/mutation_minimal/data.csv ./tmp mutation_minimal \
--config_file {unzip_path}/example/mutation_minimal/paplot.cfg
2. ヘッダなし¶
SAMPLE00,intronic,GATA3
SAMPLE00,UTR3,CDH1
SAMPLE00,exonic,GATA3
SAMPLE01,splicing,WASF3
SAMPLE01,intronic,WASF3
SAMPLE01,exonic,NRAS
SAMPLE02,intronic,FBXW7
SAMPLE02,intronic,GATA3
SAMPLE02,ncRNA_intronic,ACVR2B
SAMPLE03,exonic,CAP2
SAMPLE03,intronic,PIK3CA
SAMPLE03,downstream,SEPT12
データにヘッダ行がない場合、列名でなく列番号を設定します。 列番号は左から順に 1 始まりで数えます。
設定ファイルの [result_format_mutation] セクションでデータの列番号を次のように設定します。
[result_format_mutation]
# ヘッダオプションを False に設定
header = False
col_group = 2
col_gene = 3
col_opt_id = 1
編集した設定ファイルを使用して paplot を実行します。
paplot mutation {unzip_path}/example/mutation_noheader/data.csv ./tmp mutation_noheader \
--config_file {unzip_path}/example/mutation_noheader/paplot.cfg
3. ポップアップの情報追加¶
マウスカーソルを乗せた時に表示する情報 (ポップアップ) をカスタマイズすることができます。
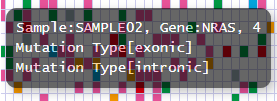
最小構成で表示するポップアップ (グリッド部分) は以下の通りサンプル、遺伝子、変異タイプが表示されています。
変更前
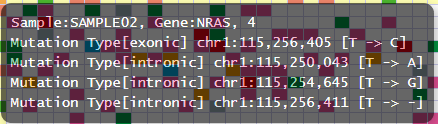
情報を追加して変異の場所と変異の内容を確認できるようにします。
変更後
Sample,Chr,Start,Ref,Alt,MutationType,Gene
SAMPLE00,chr10,8114472,A,C,intronic,GATA3
SAMPLE00,chr13,28644892,G,-,intronic,FLT3
SAMPLE00,chr13,28664636,-,G,intronic,FLT3
SAMPLE00,chr16,68795521,-,T,UTR3,CDH1
SAMPLE00,chr10,8117068,G,T,exonic,GATA3
SAMPLE00,chr3,178906688,G,A,intronic,PIK3CA
SAMPLE00,chr13,28603715,G,-,intergenic,FLT3
SAMPLE00,chr14,103368263,G,C,intronic,TRAF3
今回の例では、必須項目であるサンプル名 (Sample)、遺伝子名 (Gene)、変異タイプ (MutationType) に加えて、以下の 4 項目を追加しています。
- 染色体 (Chr)
- 変異開始位置 (Start)
- リファレンスの塩基 (Ref)
- 変異の塩基 (Alt)
まず、追加した列名を設定ファイルに記載します。
設定ファイルの [result_format_mutation] セクションでデータの列名を次のように設定します。
[result_format_mutation]
col_opt_chr = Chr
col_opt_start = Start
col_opt_ref = Ref
col_opt_alt = Alt
オプションの列名は次の形式で記述します。 col_opt_{キーワード} = {実際の列名}
次に、ポップアップの表示内容を変更します。
[mutation]
# 変更前 (最小構成の設定)
# tooltip_format_checker_partial = Mutation Type[{group}]
# 次のように変更
tooltip_format_checker_partial = Mutation Type[{group}] {chr}:{start:,} [{ref} -> {alt}]
編集した設定ファイルを使用して paplot を実行します。
paplot mutation {unzip_path}/example/mutation_option/data.csv ./tmp mutation_option \
--config_file {unzip_path}/example/mutation_option/paplot.cfg
今回はグリッド部分のポップアップを変更しました。その他のポップアップ設定項目は ポップアップの表示内容 を参照してください。
また、記載方法に関するより詳細な解説は ユーザ定義フォーマット を参照してください。