quick start¶
- paplotをインストール
- testサンプルでコマンドを実行
- 結果ファイルを表示
1. paplotをインストール¶
ここではサーバサイドでのインストールを記載しています。
ここでうまくいかない方、個人のPCにインストールする方は install を参照してください。
HGCスパコンで使用する場合、事前に
qlogin してください。git clone -b master https://github.com/Genomon-Project/paplot.git
cd paplot
python setup.py build install --user
installの確認
以下を入力してください。
pa_plot conf
このように表示されればインストール成功です。
**********************
hello paplot !!!
**********************
config file:/usr/lib/python2.7/site-packages/{paplot-versoion}-py2.7.egg/config/paplot.cfg
(このあとにデフォルト設定の内容が表示されます)
2. testサンプルでコマンドを実行¶
テストサンプルを用意していますので実行します。
cd {paplotをインストールしたディレクトリ}
# create bar graphs of qc
pa_plot qc "example/qc/*.csv" ./tmp DUMMY --config_file example/example.cfg
# create bundle graphs of Structural Variation (SV)
pa_plot sv "example/sv/*.txt" ./tmp DUMMY --config_file example/example.cfg
# create bundle graphs of Structural Variation (mutation-matrix)
pa_plot mutation example/mutation/sample_merge.csv ./tmp DUMMY --config_file example/example.cfg
3. 結果ファイルを表示¶
HTMLファイルができていますか?
{paplot をインストールしたディレクトリ}
└ tmp
├ DUMMY
│ ├ graph_mut.html <--- mutation-matrix グラフ
│ ├ graph_qc.html <--- qc グラフ
│ └ graph_ca.html <--- ca グラフ
│
├ js <--- この4つのディレクトリはHTMLファイルを表示するために必要です。消さないでください。
├ layout
├ lib
├ style
|
└ index.html <--- このファイルを web ブラウザで開いてください。
HTMLファイルを web ブラウザで開いてください。
※HGCスパコン等、サーバ上で実行した場合はファイルをローカルに転送するか、サーバ上の仮想ウィンドウ(NoMachime等)で表示してください。
ローカルに転送する場合は、
tmp ディレクトリを丸ごとコピーしてください。次のように見えていますか?
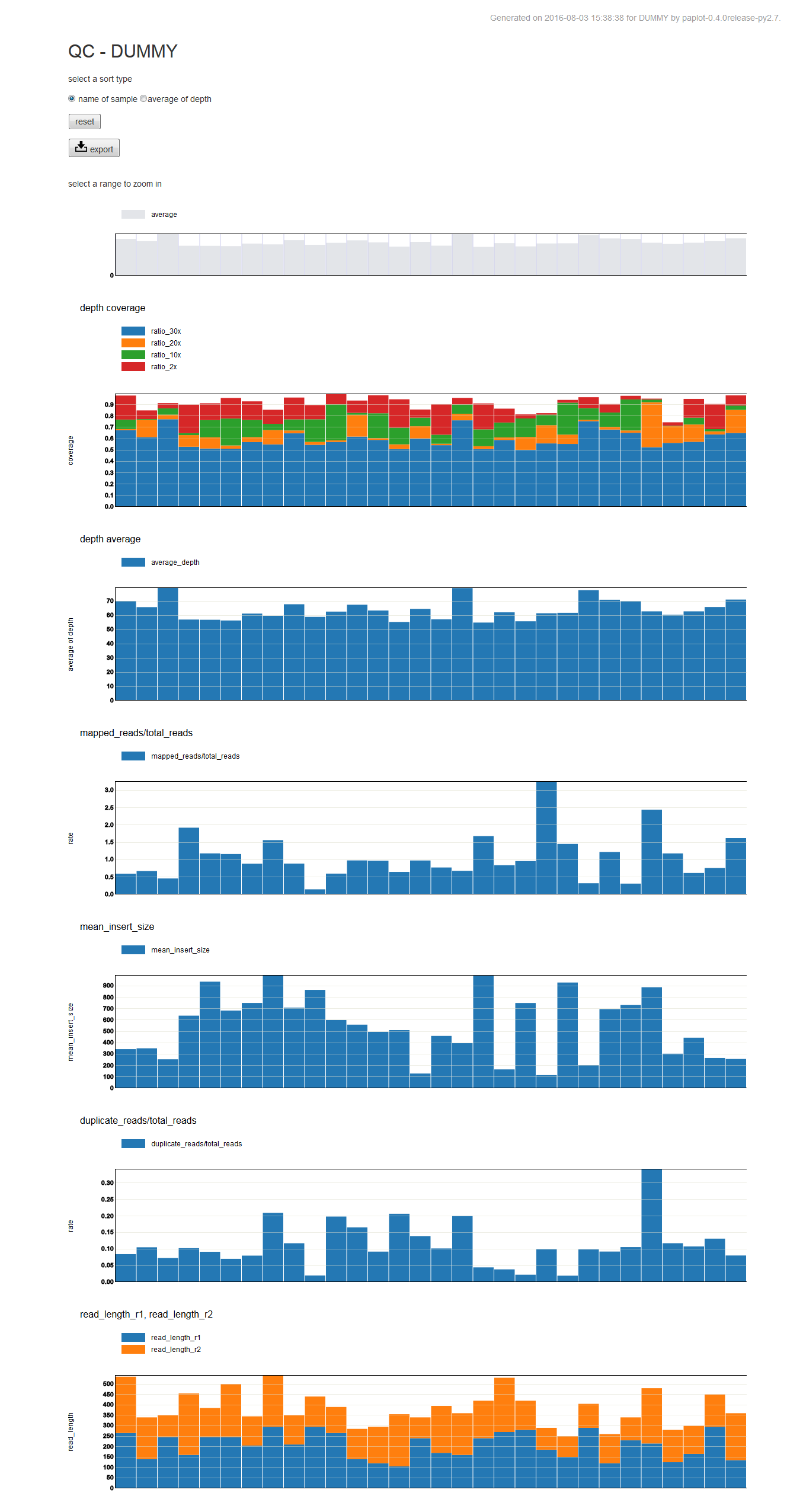
QC グラフ
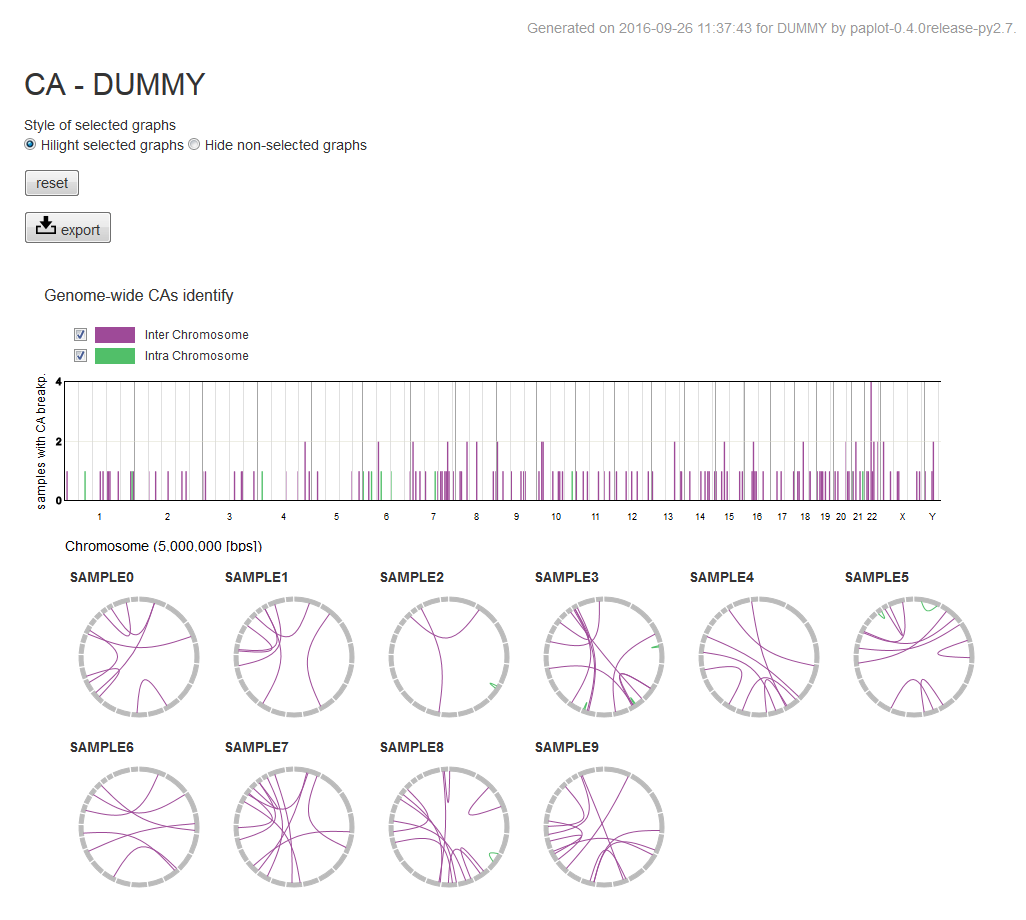
CA グラフ
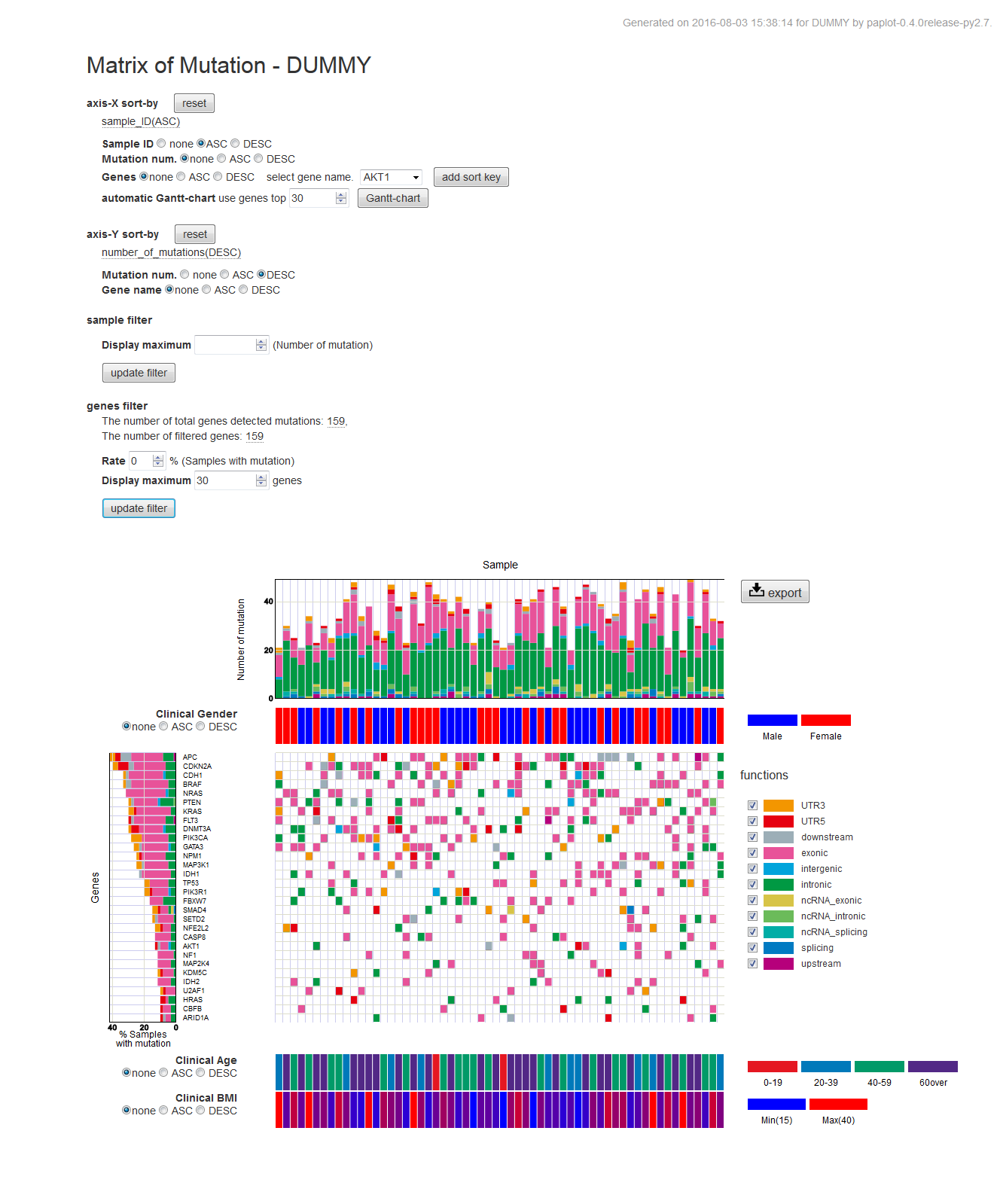
mutation-matrix グラフ
それぞれのグラフの使い方は how to use graphs を参照してください。